👁 Anotacion tipos celulares
Anotación de tipos celulares
BIENVENIDOS
Presenta: Erick Cuevas Fernández




![]()

Anotación de clusters
Algoritmo/método vs Relevancia Biológica Hay que “campechanearle”.
- Identificación de tipos y Subtipos Celulares.
- Entender la función celular.
- Descubrimiento de nuevos tipos celulares.
- Base para análisis posteriores.
Aproximaciones generales para anotar
- Modo artístico
- Usando datasets de referencia. Automático.
- Combinacion de 1 y 2.
- Anclas con Seurat
Anclas con Seurat
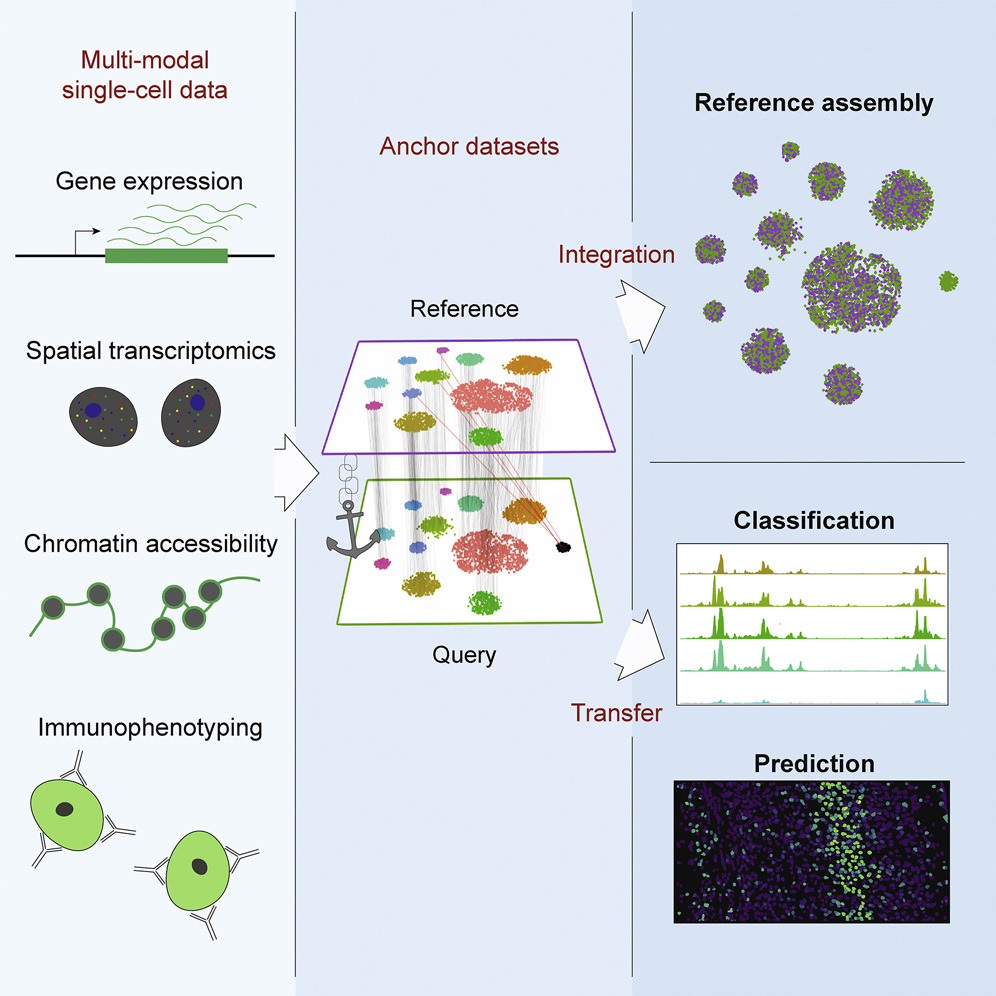
El problema con el que trabajaremos
Conjunto de datos a trabajar
La mejor estrategia para anotar, es entender el problema.

Antes de seguir, tip para aprovechar lo mejor de dos mundos SingleCellExperiment y Seurat
De Seurat a SingleCellExperiment
De SingleCellExperiment a Seurat
Notas de SeuratObject y SingleCellEXperiment Object
El objeto SCE usa la estructura de SummarizedExperiment. Los slots extra que tiene son:
- assays
- colData
- rowData
- reducedDims
El objeto Seurat se conforma de los siguientes slots:
- @data
- @meta.data
- @assays
- @reductions
Importacion de datos
Pasamos de SO a SCE
library(Seurat)
library(dplyr)
library(SingleCellExperiment)
# Recuerda descargar primero el dataset del link
so <- readRDS("alsaigh_part2.rds")
# Lo convertimos a sce
sce <- as.SingleCellExperiment(so, assay = "RNA")
colData(sce) <- as.data.frame(colData(sce)) %>%
mutate_if(is.character, as.factor) %>%
DataFrame(row.names = colnames(sce))
# Definimos resolucion
cluster_cols <- grep("res.[0-9]", colnames(colData(sce)), value = TRUE)
sapply(colData(sce)[cluster_cols], nlevels)Enfoque automático: a partir de un dataset de referencia
Dataset de referencia
# Para anotar
# cargar datos de referencia con anotaciones de Ensembl.
library(celldex)
ref.data <- HumanPrimaryCellAtlasData(ensembl=TRUE)
library(biomaRt)
library(SummarizedExperiment)
# Seleccione la base de datos Ensembl
ensembl <- useEnsembl(biomart = "ensembl", dataset = "hsapiens_gene_ensembl")
# Extraer los ID de Ensembl del objeto SummarizedExperiment
ensembl_ids <- rownames(ref.data)
# Obtener los símbolos genéticos correspondientes a los ID de Ensembl
annotations <- getBM(attributes = c("ensembl_gene_id", "hgnc_symbol"),
filters = "ensembl_gene_id", values = ensembl_ids, mart = ensembl)
# Fusionar anotaciones con rowData del objeto SummarizedExperiment
rowData(ref.data)$geneSymbol <- annotations$hgnc_symbol[match(ensembl_ids, annotations$ensembl_gene_id)]
rownames(ref.data) <- rowData(ref.data)$geneSymbolUsando SingleR
Usando SingleR
library(SingleR)
library(scater)
pred <- SingleR(test = sce,
ref = ref.data,
labels = ref.data$label.main,
assay.type.test=1,
BPPARAM= BiocParallel::MulticoreParam(6)) # 6 CPUs.
table(pred$labels)Parámetros Principales:
- test: Una matriz de expresión donde las filas son genes y las columnas son células a anotar.
- ref: Una lista de matrices de expresión que representan las bases de datos de referencia.
- labels: Etiquetas para los tipos celulares en las bases de datos de referencia.
- de.method: Método para calcular los genes diferencialmente expresados.
- BPPARAM: Parámetros para la paralelización.
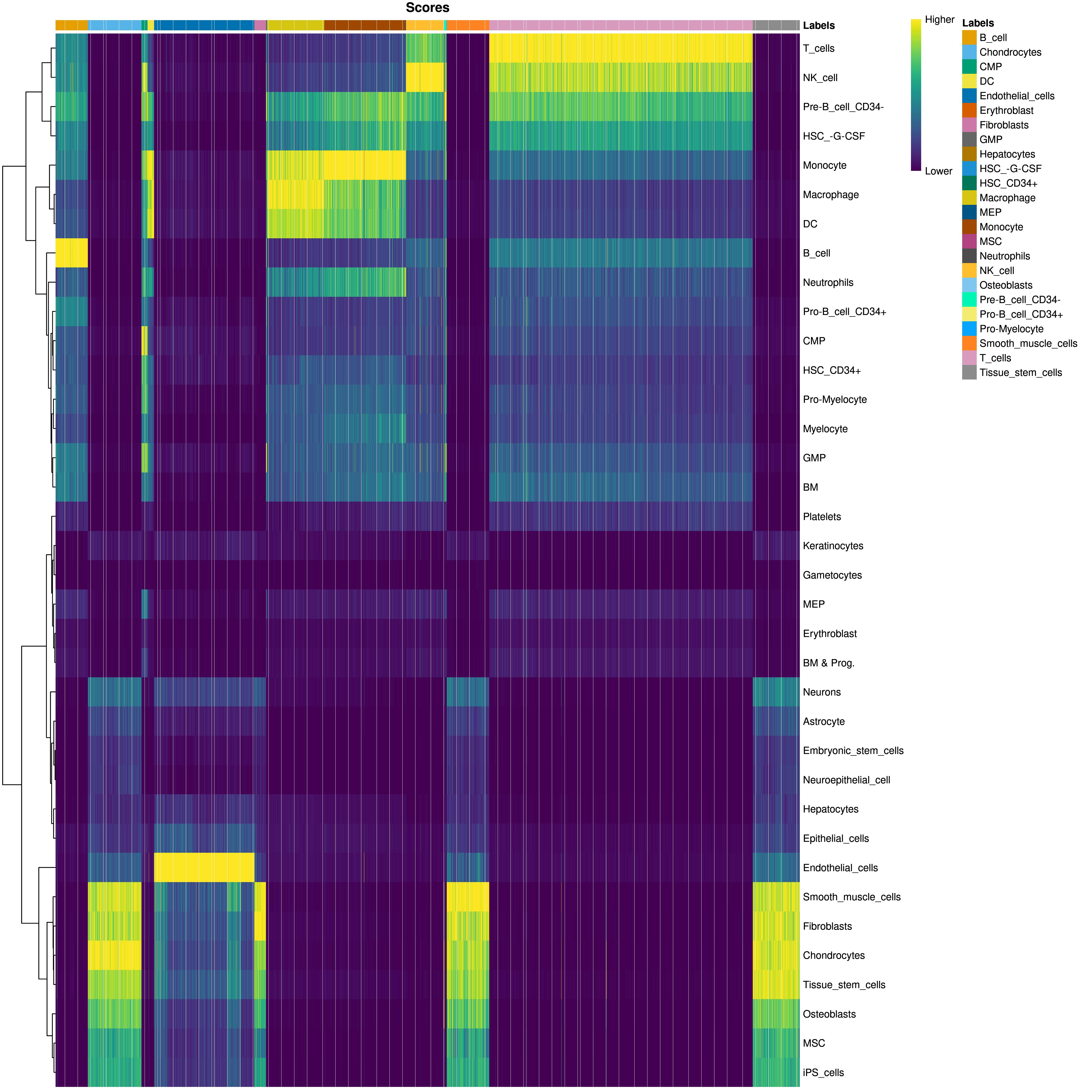
Observar calidad de predicción
summary(is.na(pred$pruned.labels))
# Grafico para ver las celulas eliminadas
# plotDeltaDistribution(pred.grun, ncol = 3)
to.remove <- is.na(pred$pruned.labels)
table(Label=pred$labels, Removed=to.remove)
to.remove <- pruneScores(pred, min.diff.med=0.2)
table(Label=pred$labels, Removed=to.remove)
# Modificamos nuestro sce
sce_dummy <- sce[,to.remove]
sce_dummy$new_labels <- pred$labels[to.remove]
table(sce_dummy$new_labels)
plotReducedDim(sce_dummy, "TSNE", colour_by="new_labels")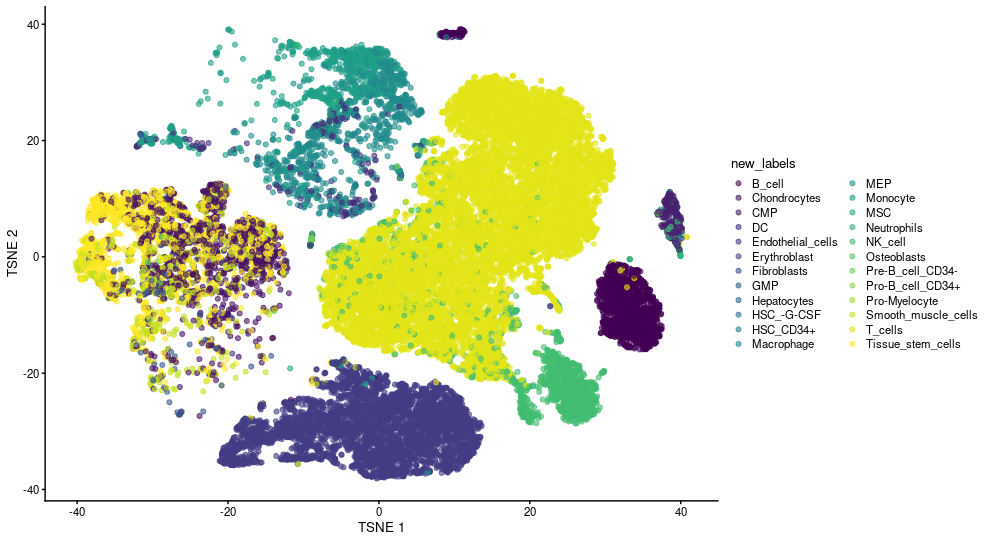
Aproximación “artesanal”
¿Qué hacemos si nuestra anotación aún no es la “ideal”?

Podemos primero eliminar
Ver que tipos celulares son “innecesarios”
library(Seurat)
# Recuerda que el archivo original era un "so"
so@meta.data$Proplabels <- sce$ProbLabels
# Le transferimos las etiquetas al objeto Seurat
Idents(so) <- so$Proplabels
table(Idents(so))
# Eliminamos los tipos celulares con menos de 350 OJO solo para este ejemplo
`%!in%` <- Negate(`%in%`)
so <- subset(so, (Proplabels %!in% c("Pro-Myelocyte", "MEP", "Pro-B_cell_CD34+", "Pro-B_cell_CD34+", "HSC_-G-CSF", "Hepatocytes", "MSC","Osteoblasts","HSC_CD34+","Erythroblast", "Pre-B_cell_CD34-","GMP","Neutrophils","CMP","DC")))
table(Idents(so))
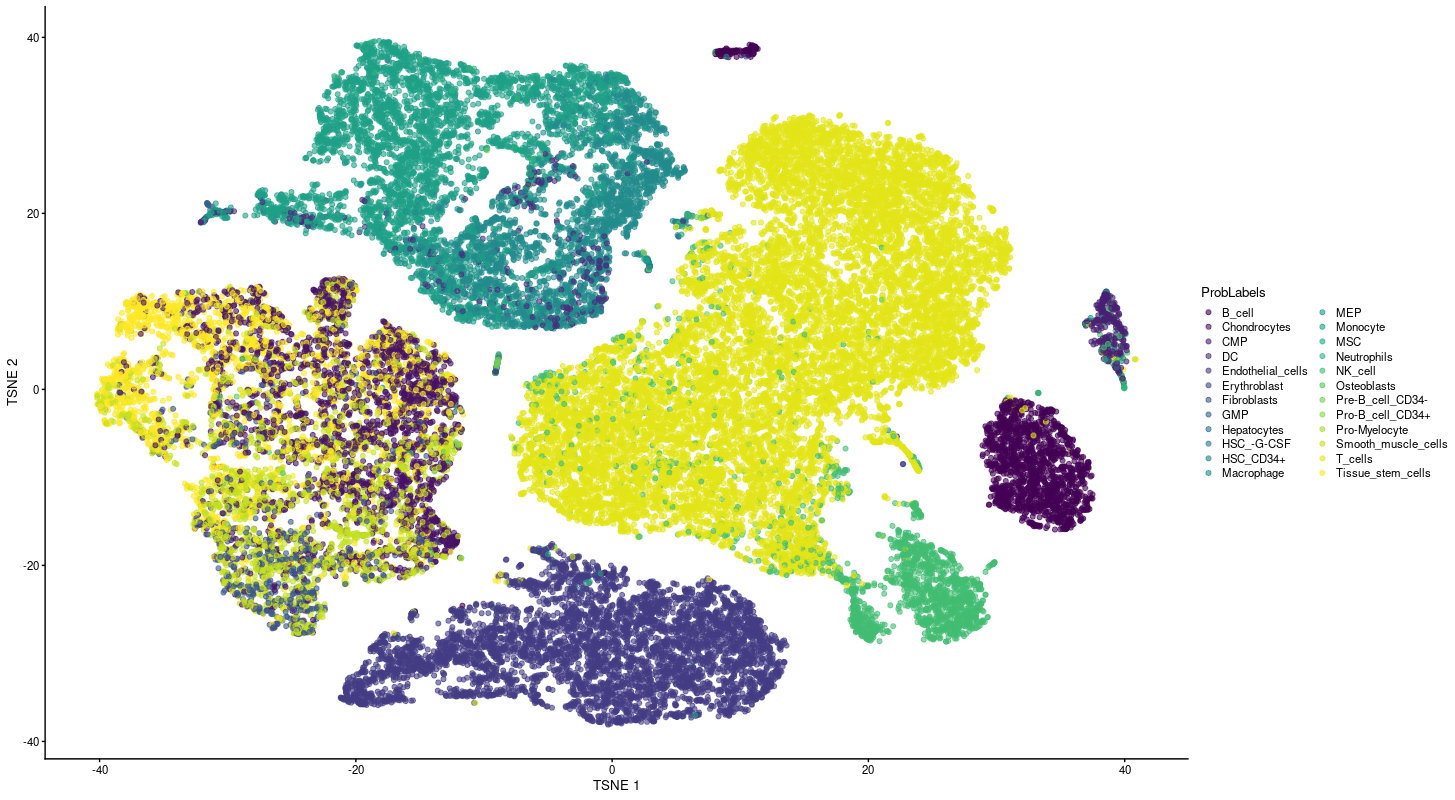
# Observamos nuestros clusters
DimPlot(so, reduction = "tsne")Ahora se ven asi nuestro clusters
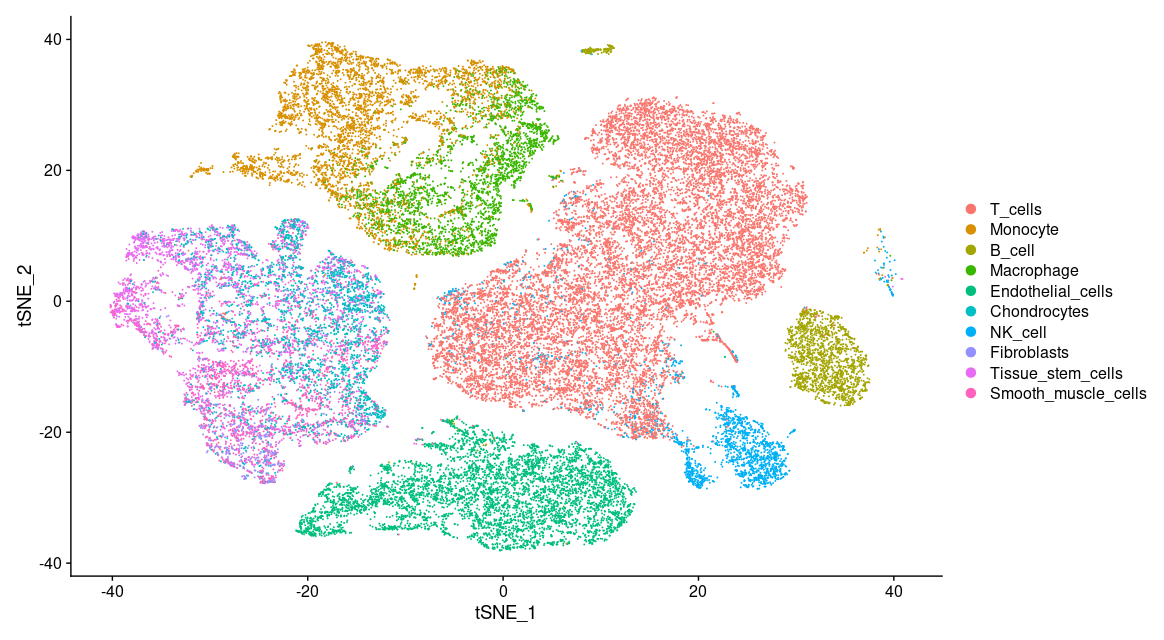
¿Y si quiero agregar un tipo celular basado en genes marcadores?

Observar los marcadores
Y de este modo se va dando una identidad a cada cluster.
# Marcadores ya reportados para celulas espumosas
# so <- readRDS( "alsaigh_PC20_part3.rds")
foamy_cells <- c("FABP5", "GPNMB", "TREM2", "APOC1", "CD9", "SPP1")
# so <- ScaleData(so, display.progress = FALSE)
cell_typeA_marker_gene_list <- list(c("FABP5", "GPNMB", "TREM2", "APOC1", "CD9", "SPP1"))
DefaultAssay(so) <- "RNA"
so <- AddModuleScore(object = so,
features = cell_typeA_marker_gene_list,
# ctrl = 1,
name = "espumosas")
FeaturePlot(object = so_new, features = "espumosas1", reduction = "tsne")Observamos la expresion en conjunto
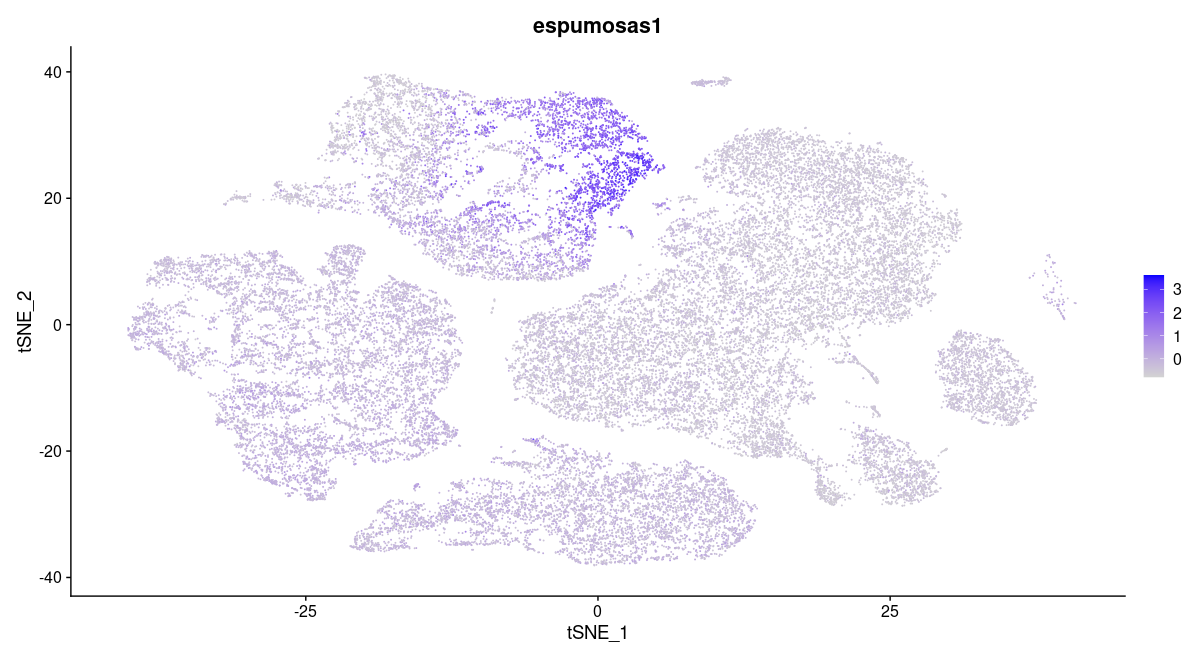
Cambiamos el nombre de nuestras celulas
select_foamy <- colnames(so)[so$espumosas1 > 1.5]
new_cells <- so$Proplabels
# Hacemos un loop para sustituir el nombre de las etiquetas
for(i in 1:length(colnames(so) %in% select_foamy)){
if((colnames(so) %in% select_foamy)[i]){
new_cells[i] <- "Foamy_cells"
} else{
new_cells[i] <- so$Proplabels[i]
}
}
so$new_cells <- new_cells
Idents(so) <- so$new_cells
DimPlot(so, reduction = "tsne")
DimPlot(so, reduction = "umap")
DimPlot(so, reduction = "umap", group.by = "region")
DimPlot(so, reduction = "umap", group.by = "sample_id")tSNE
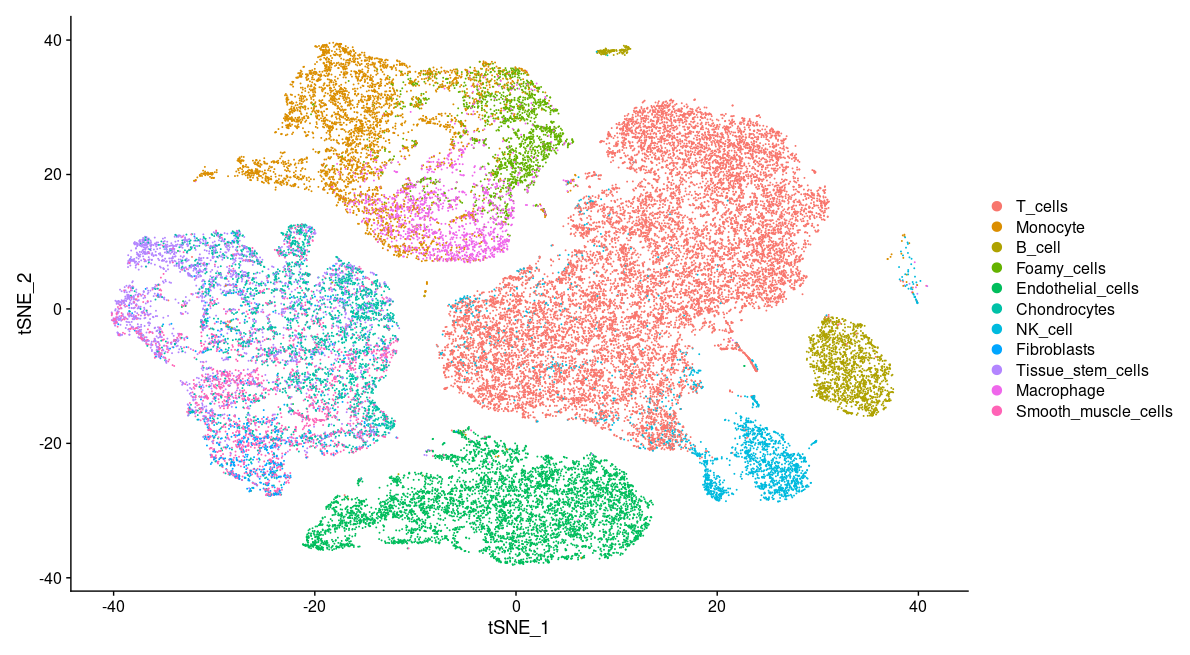
UMAP
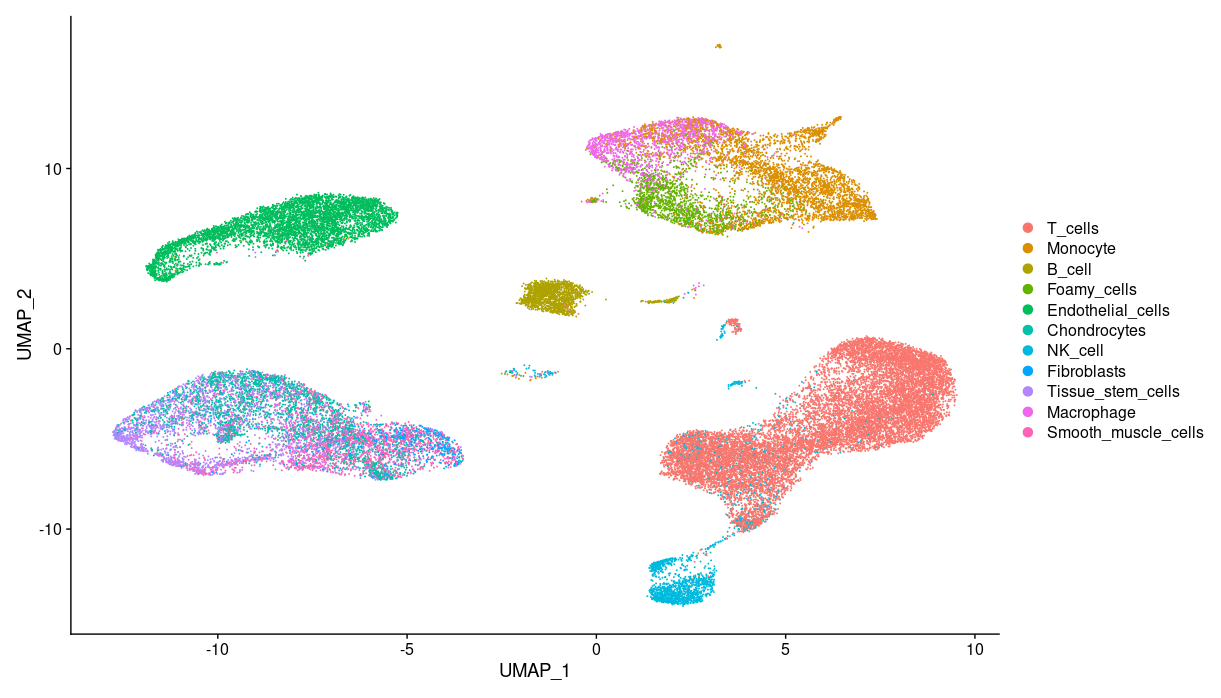
¿Tiene sentido?
df_cells_cts <- table(Idents(so), so$region) %>% as.data.frame()
colnames(df_cells_cts) <- c("cell_type", "region", "freq")
df_cells_cts <- df_cells_cts %>%
group_by(cell_type) %>%
mutate(prop = freq / sum(freq))
ggplot(df_cells_cts, aes(x = cell_type, y = prop, fill = region)) +
geom_bar(stat = "identity", position = position_dodge(), color = "black") +
theme_classic() +
labs(x = "Cell Type",
y = "Proportion",
fill = "Femoral Artery Plaque") +
scale_y_continuous(labels = scales::percent, limits = c(0, 1)) +
theme(axis.text.x = element_text(angle = 45, hjust = 1),
text = element_text(size = 15))
# Encontrar "nuevos" marcadores
# Cuidado con tu memoria RAM!
f.markers <- FindAllMarkers(so,
only.pos = FALSE)
f.markers %>%
group_by(cluster) %>%
top_n(n = 10, wt = avg_log2FC) -> top10
#so <- ScaleData(so)
DoHeatmap(so, features = top10$gene)¿Tiene sentido?
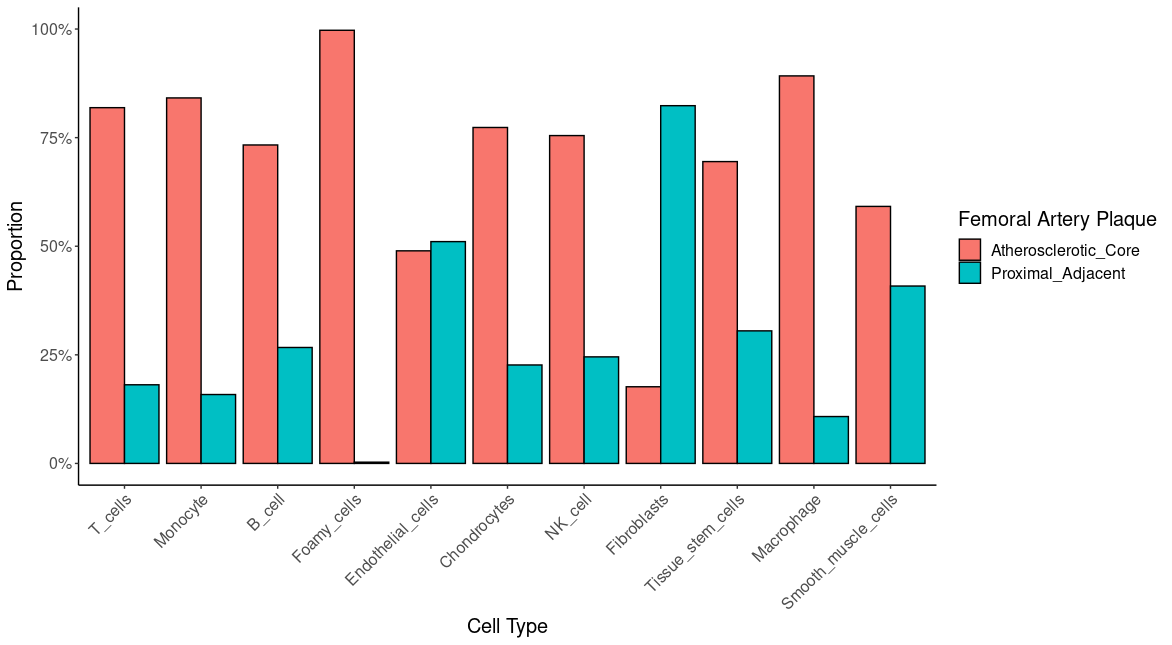
¿Tiene sentido?
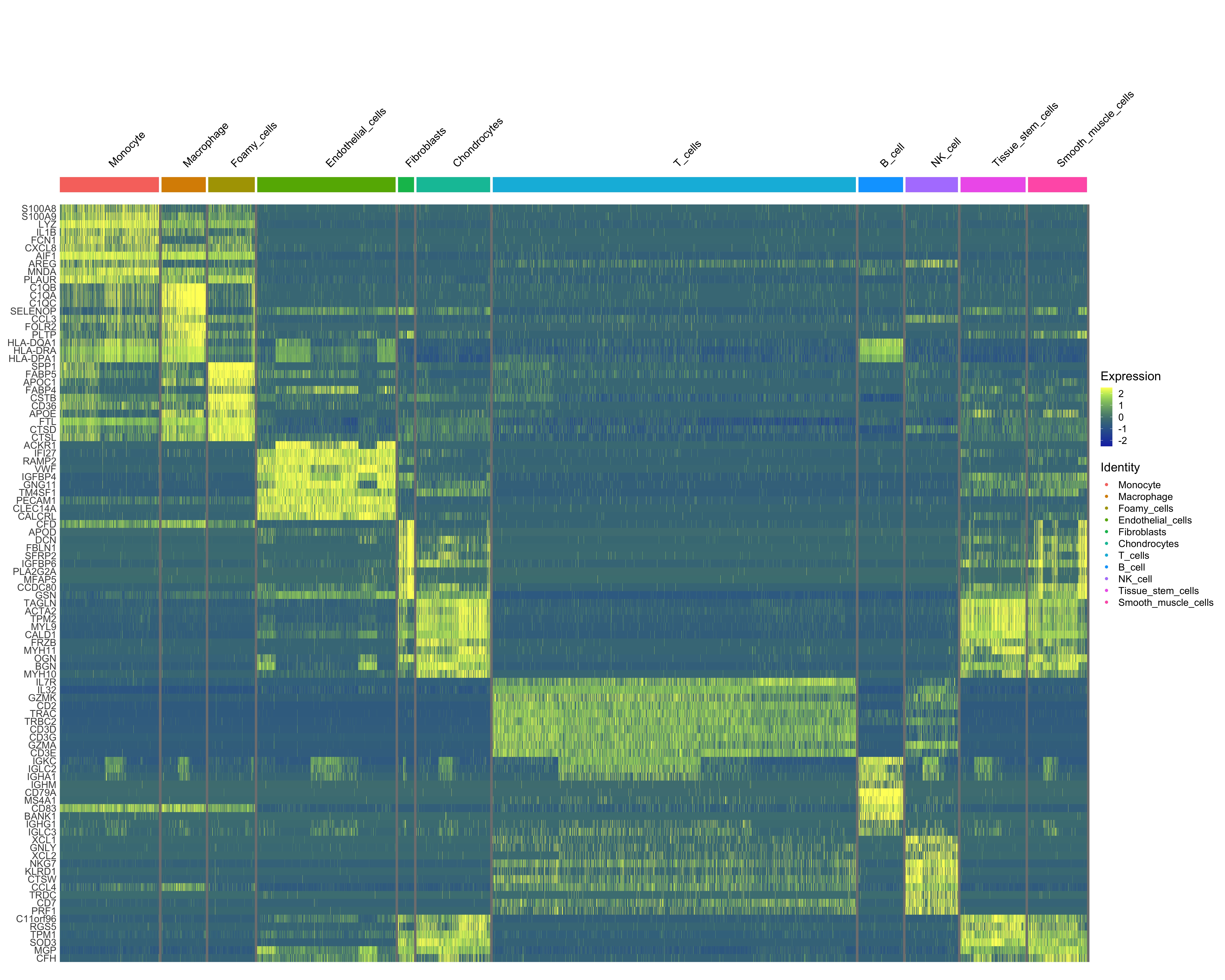
Lo que sigue…
- Expresión diferencial: por condición, región, tejido, entre tipos celulares, sexo, etc.
- Análisis de trayectoria: monocle3.
El fin
MUCHAS GRACIAS


EBM 2023